PicoSeq : le séquençage génétique sur molécule unique à portée de main
Vincent Croquette, David Bensimon, directeurs de recherche CNRS, Jean-François Allemand, professeur ENS, et leur équipe, au Laboratoire de physique statistique de l’Ecole Normale Supérieure (CNRS / ENS / UPMC / Université Paris Diderot), à Paris, sont connus pour leur technique de manipulation d’une molécule d’ADN unique, à l’aide d’un piège magnétique. Il y a trois ans, raffinant leur protocole à l’extrême, ces physiciens ont réalisé que ce dernier offrait une méthode originale de décryptage du génome. A la clé, la création de PicoSeq, une start-up déjà bardée de prix qui n’attend plus qu’une levée de fonds pour pouvoir proposer une version commercialisable de son innovation.
Car, à l’origine, rien de prédestinait ce groupe de physiciens à s’engager sur le chemin de la création d’entreprise. Leur spécialité : un piège magnétique qui permet d’appliquer une force mécanique à une longue molécule afin d’étudier ses propriétés. Grâce à quoi, ces physiciens sont notamment parvenus à étudier en détails les mécanismes d’enroulement et d’étirement de l’ADN impliqués dans sa réplication.
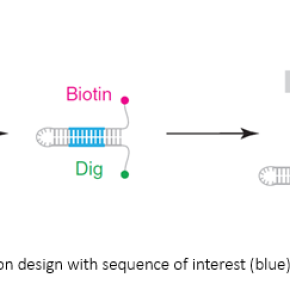
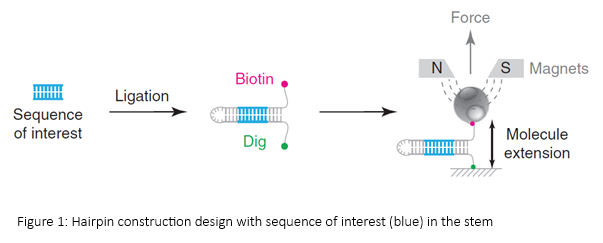
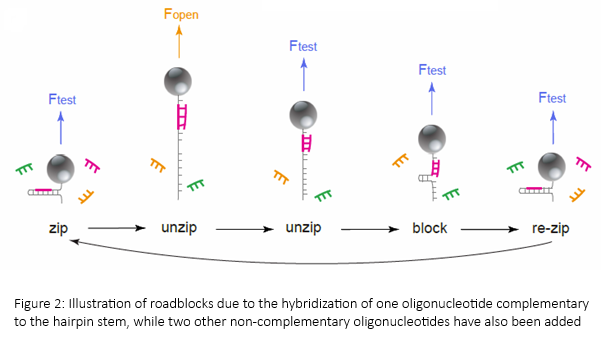
Mais en 2011, Vincent Croquette réalise que grâce à l’utilisation de petits fragments d’ADN dont la séquence est connue à l’avance, les oligonucléotides, il peut également lire l’information génétique contenue sur une molécule d’ADN quelconque, une fois ses deux brins séparés par le piège magnétique. « Il devenait alors possible de faire le séquençage d’une molécule unique, sans recours aux techniques d’amplification utilisées par les méthodes actuelles et sans recours à des produits fluorescents chers », précise le scientifique.
Ainsi, plusieurs brevets sont déposés en 2011 et PicoSeq fondée l’année suivante. S’en suivent quatre prix dont celui du concours mondial de l’innovation, décerné au début de l’année 2014. Objectif actuel : le développement de tests de dépistage du syndrome de l’X fragile, associé à la moitié des cas de retard mental. « Cette maladie est causée par une répétition plusieurs centaines de fois de la séquence GGC en amont du gène FMR1 sur le chromosome X. Or les méthodes traditionnelles de séquençage peinent à déterminer précisément le nombre de ces répétitions du fait d’erreurs de recopiage lors de la phase d’amplification », explique Vincent Croquette. Il ajoute : « Nous ne sommes plus très loin d’apporter la preuve de principe de l’efficacité de notre méthode dans ce cas précis ». Après quoi, la balle sera clairement dans le camp des investisseurs. De quoi transformer PicoSeq en un acteur incontournable du séquençage génétique !
En savoir plus
PicoSeq, la start-up qui revoit le séquençage d’ADN (L’Express L’Entreprise) - mars 2013