Comment s’assemble un virus icosaédrique ?
Une équipe de chercheurs a suivi la reconstitution spontanée d’un virus de plante grâce à la technique dite de diffusion des rayons X aux petits angles. Ils ont ainsi pu observer le génome agir comme un support d’assemblage pour les sous-unités protéiques formant la coque du virus.
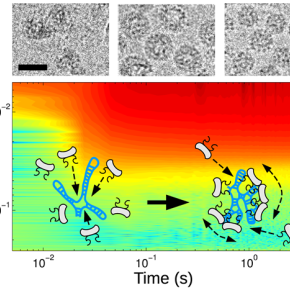
Le virus de la marbrure chlorotique de la cornille (CCMV en anglais) est un virus à ARN infectant une variété de haricots. Quatre segments d’ARN encodant le génome viral sont répartis à l’intérieur de trois capsides distinctes nécessaires à la réplication du virus. Ces capsides, qui protègent l’ARN, sont constituées de 90 sous-unités protéiques arrangées en une structure formant un solide à 20 faces, un icosaèdre. La survie du virus repose en partie sur sa capacité à s’assembler rapidement, et sans défaut, au sein de la cellule hôte. Très peu de mesures expérimentales sont à ce jour disponibles du fait de la difficulté à détecter des molécules biologiques sur une large gamme d’échelle de temps.
Pour la première fois, une équipe du Laboratoire de physique des solides (LPS, CNRS/Univ. Paris-Sud), impliquant également des chercheurs de l’Institut de biologie intégrative de la cellule (I2BC, CNRS/CEA/Univ. Paris-Sud), du Laboratoire Léon Brillouin (LLB, CNRS/CEA) et de l’European Synchrotron Radiation Facility, est parvenue à élucider la dynamique d’assemblage de ce virus.
Le virus a été reconstitué à partir de sous-unités protéiques et d’ARN génomique purifiés, et son assemblage spontané a été suivi par la méthode de diffusion des rayons X aux petits angles, résolue en temps avec une source synchrotron. Une capside vide s’assemble séquentiellement et nécessite un positionnement précis des sous-unités au cours du processus. Pour un virus complet, en revanche, le génome agit comme un support d’assemblage et capture en moins d’une seconde un grand nombre de sous-unités pour former un complexe désordonné. Ce dernier s’auto-organise sur des temps longs tout en poursuivant la capture des sous-unités manquantes, et donne lieu à la capside protectrice. Ce mécanisme serait avantageux pour à la fois associer rapidement les composants élémentaires du virus, et pour assurer la sélectivité du génome et corriger les éventuelles erreurs d’assemblage.
La compréhension de tels mécanismes pourrait permettre la mise au point de techniques thérapeutiques afin d’éviter la réplication de ces virus.
Référence
Nonequilibrium self-assembly dynamics of icosahedral viral capsids packaging genome or polyelectrolyte
M. Chevreuil, D. Law-Hine, J. Chen, S. Bressanelli, S. Combet, D. Constentin, J.Degrouard, J. Möller, M. Zeghal et G. Tresset
Nature Communication 9 (2018), Article number : 3071 doi:10.1038/s41467-018-05426-8